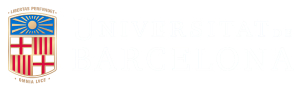Eines de bioinformàtica per a la interpretació biològica i la visualització de dades
Des del grup hem desenvolupat eines com la Food-Biomarker Ontology (FOBI) (Castellano-Escuder P, et al., 2020), la primera ontologia dissenyada per integrar dades de metabolòmica i nutrició, i POMAShiny (Castellano-Escuder P, et al., 2021), que ofereix mètodes estadístics univariants, mètodes multivariants i de reducció de dimensions, tècniques de selecció de característiques, enfocaments d’anàlisi de regressió regularitzada, algorismes de classificació basats en aprenentatge automàtic, estratègies de models de predicció i diverses opcions d’interacció visual d’alta qualitat.
Seguint els principis FAIR, ambdòs codis font i els fitxers de dades estan disponibles a partir dels repositoris públics de GitHub.
The Food-Biomarker Ontology (FOBI) és la primera ontologia creada per integrar dades de metabolòmica i nutrició (Castellano-Escuder P, et al., 2020). Aquesta ontologia té com a objectiu relacionar diferents tipus d’aliments amb els seus metabòlits associats o biomarcadors d’ingesta alimentària.
FOBI consta de 1.197 termes, 4 propietats diferents, 13 classes d’aliments de nivell superior, 11 classes de biomarcadors de nivell superior i més de 4.500 relacions. A més, FOBI forma part del projecte OBOFoundry, i els identificadors de FOBI s’han indexat a les bases de dades HMDB i FooDB per facilitar la interoperabilitat i l’intercanvi de dades.

Food-Biomarker Ontology (FOBI)
Arquitectura de FOBI considerant la poma com a exemple.
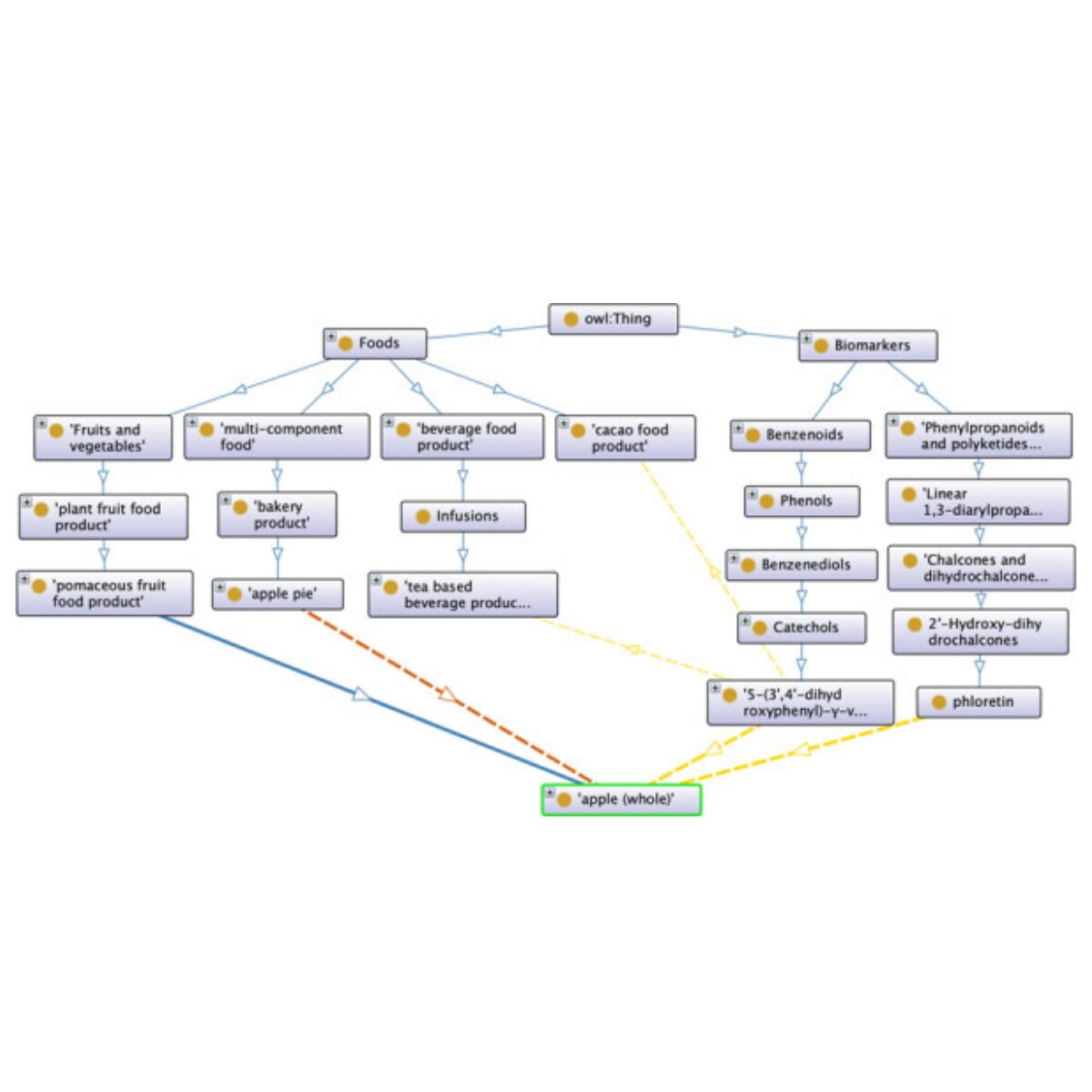
- Anàlisi d’enriquiment de classes químiques: ORA i MSEA utilitzant les classes químiques de FOBI com a conjunts de metabòlits.
- Anàlisi d’enriquiment d’aliments: ORA i MSEA utilitzant els grups d’aliments de FOBI com a conjunts de metabòlits.

POMAShiny
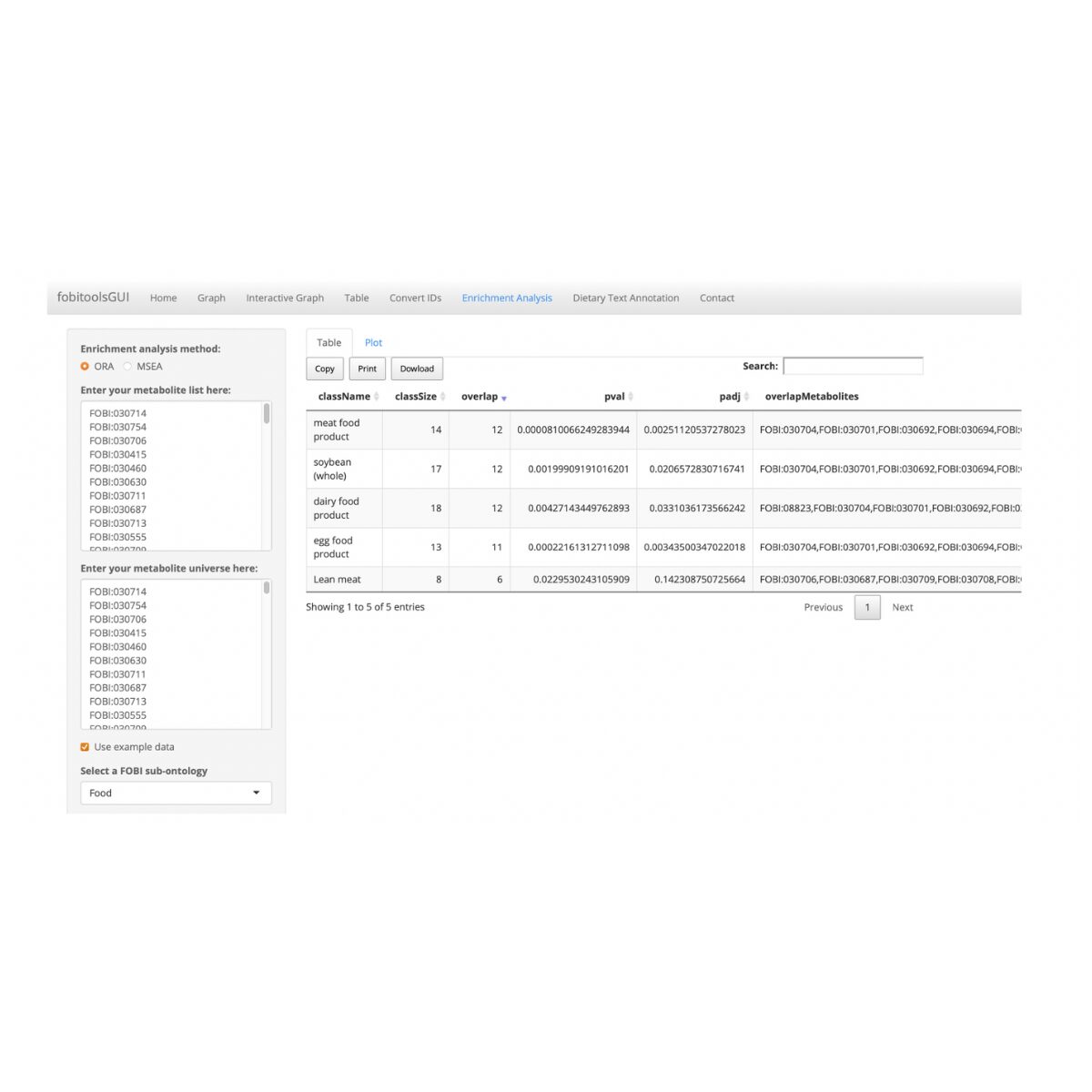
POMAShiny és una eina web que ofereix un flux de treball estructurat, flexible i fàcil d’utilitzar per al processament, exploració i anàlisi estadística de dades metabolòmiques. Aquesta eina es basa en el paquet POMA de R/Bioconductor, que augmenta la reproductibilitat i la flexibilitat de l’anàlisi fora de l’entorn web. El flux de treball de POMAShiny està estructurat en quatre panells seqüencials i ben definits:
- Carrega de dades,
- Preprocessament,
- EDA (Exploració de Dades),
- Anàlisi estadística.
POMAShiny
POMAShiny requereix dos fitxers en format CSV com a entrada: un fitxer de metadades (target) i un fitxer de característiques (features). El fitxer de metadades ha d’incloure els noms de les mostres a la primera columna, les etiquetes de grup (per exemple, control i cas) a la segona, i opcionalment, covariables rellevants a partir de la tercera columna. El fitxer de característiques conté les característiques quantificades de l’experiment, amb una característica per columna. L’ordre de les files ha de ser el mateix en ambdós fitxers. Un cop carregats, POMAShiny converteix els fitxers en un objecte MSnSet, segons el paquet MSnbase de R/Bioconductor.
Els usuaris poden seleccionar mostres específiques al fitxer de metadades per crear subconjunts de dades per a l’anàlisi. A més, POMAShiny ofereix la funció opcional de combinar característiques que pertanyen a una mateixa entitat (com pèptids d’una proteïna o ions d’un compost). Per utilitzar aquesta funció, cal un fitxer de “grup” (CSV) que indiqui quines característiques s’han de combinar. També permet descarregar una taula amb els coeficients de variació de les característiques combinades.
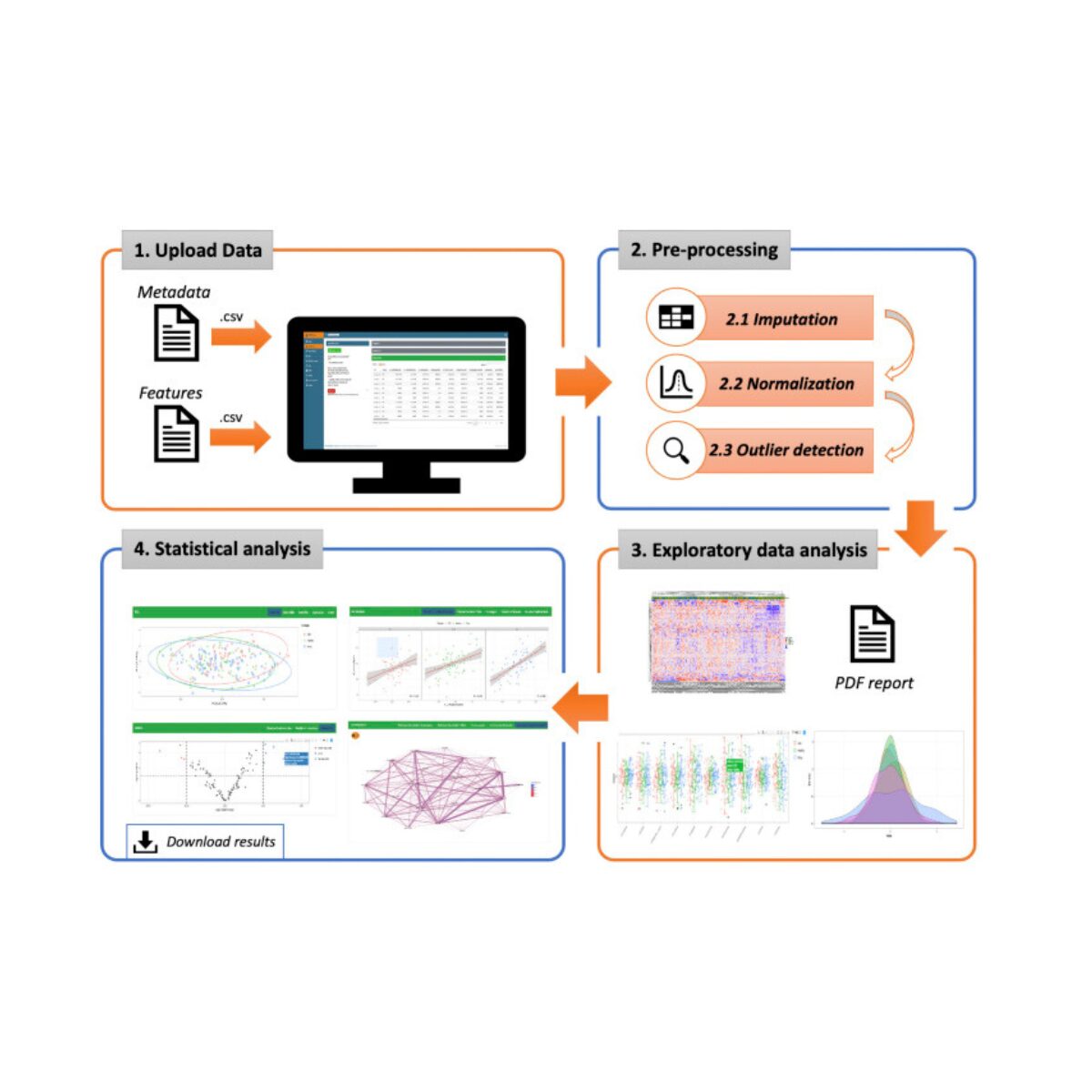

L’EDA ajuda a identificar factors incontrolats i possibles valors anòmals, i és recomanable realitzar-la abans de l’anàlisi estadística. A més, en absència de biaixos significatius, l’EDA pot proporcionar una visió inicial de les característiques més rellevants de l’estudi.
POMAShiny ofereix gràfics interactius i personalitzables per a l’EDA, incloent-hi gràfics de volcà (per a estudis de dos grups), boxplots, gràfics de densitat i mapes de calor agrupats. També inclou opcions per a anàlisi de components principals (PCA) i anàlisi de clústers.

Aquest panell inclou diversos mètodes estadístics, des dels enfocaments més habituals en l’anàlisi de dades de metabolòmica i proteòmica fins a altres metodologies menys freqüents en aquests camps. Tots els mètodes estadístics que ofereix POMAShiny estan implementats de manera molt intuïtiva per a l’usuari i generen tant taules descarregables com gràfics interactius com a resultats. Aquests anàlisis són els següents:
- Anàlisi univariant
- Limma
- Anàlisi multivariant
- Anàlisi de clústers
- Anàlisi de correlació
- Regressió regularitzada
- Boscos aleatoris
- Ràtio d’odds
- Productes de rang